Determination of the Methylation Status of RCGY Sites in the Human Genome: A Comparison of NGS Analysis of GlaI DNA Hydrolysates and GlaI-PCR Data
M.A. Abdurashitov, A.G. Akishev, S.Kh. Degtyarev[]
M.A. Abdurashitov, A.G. Akishev, S.Kh. Degtyarev
SibEnzyme Ltd, Novosibirsk
Abstract
The 5-methylcytosine-dependent DNA endonuclease GlaI recognizes and cleaves DNA sites with the sequence 5′-R(5mC)GY-3′ / 3′-YG(5mC)R-5′, which are formed de novo in the human genome by the DNA methyltransferase DNMT3. Owing to this specificity, GlaI is efficiently used to determine the methylation status of RCGY sites in the human genome. Genome-wide mapping of R(5mC)GY sites can be performed by next-generation sequencing (NGS) of DNA hydrolysates generated by GlaI digestion, whereas analysis of individual DNA fragments can be carried out using GlaI-PCR analysis.
In this study, we compared the results of NGS analysis of GlaI DNA hydrolysates with data obtained by GlaI-PCR analysis of regulatory regions of 11 tumor suppressor genes in DNA from human cell lines L68 (normal lung fibroblasts), Raji (Burkitt lymphoma), and U937 (histiocytic lymphoma). It was shown that in L68 cells most of the analyzed regulatory regions are unmethylated, whereas methylation of these regions is observed in the malignant cell lines Raji and U937. With the exception of a single region, for all other analyzed fragments the methylation status determined by GlaI-PCR analysis fully correlates with the number of R(5mC)GY sites detected by NGS.
The only discrepancy between the two methods was identified for the regulatory region of the RASSF1A gene in L68 cells and was attributed to the presence of allele-specific methylation, which was confirmed by additional real-time GlaI-PCR analysis. Taking allele-specific methylation of the RASSF1A regulatory region into account, complete concordance was observed between the data obtained by NGS analysis of GlaI DNA hydrolysates and GlaI-PCR analysis of regulatory regions of the studied tumor suppressor genes in the human cell lines L68, Raji, and U937.
Thus, NGS analysis of GlaI DNA hydrolysates and GlaI-PCR analysis are complementary approaches that enable reliable identification of aberrantly methylated genomic regions in normal and malignant cells, making them promising tools for epigenetic research and DNA-based diagnostics.
Keywords: 5-methylcytosine; DNA methylation; methyl-dependent restriction endonucleases;
GlaI; NGS analysis; GlaI-PCR analysis; epigenetic DNA diagnostics; allele-specific methylation.
DOI: 10.26213/3034-4298.2025.6.1.001
Citation: M.A. Abdurashitov, A.G. Akishev, S.Kh. Degtyarev (2025) Determination of the Methylation Status of RCGY Sites in the Human Genome: A Comparison of NGS Analysis of GlaI DNA Hydrolysates and GlaI-PCR Data, Epigenetic DNA diagnostics, vol 2025(1), DOI: 10.26213/3034-4298.2025.6.1.001
![]()
This work is available at Creative Commons Attribution-NonCommercial-NoDerivatives 4.0 International
Введение
5-метилцитозин-зависимая ДНК-эндонуклеаза GlaI узнает и расщепляет в ДНК человека сайты 5’-R(5mC)GY-3’/3’-YG(5mC)R-5’ [1]. При этом, метилирование ДНК de novo в геноме человека и млекопитающих осуществляется ДНК-метилазой DNMT3 с образованием именно сайтов 5’-R(5mC)GY-3’/3’-YG(5mC)R-5’. Вследствие этого, GlaI может быть использован для определения статуса метилирования сайтов RCGY в геноме человека и млекопитающих. Определение сайтов R(5mC)GY в геноме человека (эпигеномное сиквенирование) проводится путем NGS GlaI-гидролизатов ДНК человека [2].
Ранее нами было установлено, что в NGS GlaI-гидролизатах ДНК клеточных линий человека (малигнантных Raji и U937 и контрольной – клетки фибробластов легких – L68) при использовании Genome Analyzer IIx (Illumina, США) выявляется около 1,7 млн достоверно метилированных сайтов RCGY (каждый из которых представлен не менее 7-ми ридами в прочтении генома) [3], что составляет приблизительно четверть всех сайтов RCGY (7,3 млн), присутствующих в референсном геноме человека (GRCh38/hg38, [4]).
Известно, что от 20% до 40% всех CG-динуклеотидов в геноме человека не метилированы [5]. Таким образом, секвенирование GlaI-гидролизатов ДНК человека выявляет приблизительно 30-40% всех сайтов R(5mC)GY, присутствующих в геноме человека.
С целью определения статуса метилирования фрагмента ДНК или конкретного сайта RCGY в геномной ДНК можно использовать GlaI-ПЦР анализ, который заключается в обработке ДНК метилзависимой эндонуклеазой GlaI и последующей ПЦР с праймеров, окаймляющих исследуемый фрагмент ДНК с сайтами RCGY [6]. Ранее, метод GlaI-ПЦР анализа был использован для определения статуса метилирования регуляторных областей ряда генов-онкосупрессоров в ДНК клеточных линий Raji, U937 и L68 [6-7].
В настоящей работе мы провели сравнение результатов NGS GlaI-гидролизатов ДНК клеточных линий человека и данных GlaI-ПЦР анализа.
Материалы и Методы
Геномная ДНК из линий клеток человека (L-68, Raji и U-937), деионизованная вода, 1х буфер для хранения и разбавления ферментов (10 мМ Tris-HCl pH 7.6 при 25°С; 50 mM KCl; 0,1 mM EDTA; 200 µg/ml BSA; 1 mM DTT; 50% глицерин), БСА, 10x TМN буфер (100 mM Tris-HCl pH 7.9 at 25 °C; 50 mM MgCl2; 250 mM NaCl), 50 mM MgCl2, смесь dNTP (в концентрации 10 mM каждый), 5x SE стабилизатор PCR (2.7 M betaine, 6.7 mM DTT, 6.7% of DMSO), 10x SE AS буфер (670 mM Tris-HCl (pH 8.8 при 25°C); 166 mM (NH4)2SO4; 0.1% Tween-20), дезоксиолигонуклеотиды, набор для GlaI-ПЦР анализа, ДНК-маркер “100 bp”, метилзависимая сайт-специфическая эндонуклеаза GlaI и HotStart Taq-ДНК полимераза – производства ООО «СибЭнзайм» (Новосибирск, Россия). Флуоресцентно-меченный зонд производства компании «Синтол» (Москва).
Гидролиз геномной ДНК проводили как описано в инструкции к набору для проведения GlaI-ПЦР анализа, за исключением того, что в случае фрагмента RASSF1 (804пары оснований) вместо 20 нг брали 40 нг ДНК.
ПЦР проводили c использованием набора для GC-ПЦР и HotStart Taq ДНК-полимеразы на термоциклерах Терцик («ДНК-технология», Россия) для DAPK1 , RASSF2, SFRP1, HS3ST2, TWIST1, RASSF1(804) и на CFX 96 (Bio-Rad, США) – для RASSF1(173).
Для расчета специфических праймеров и зондов использовали нуклеотидные последовательности из базы данных GenBank (http://ncbi.nlm.nih.gov/genbank) по версии генома человека GRCh38/hg38, семейство программ “Vector NTI 11.5” (Invitrogen, США) и онлайн-ресурс «BLAST» (http://blast.ncbi.nlm.nih.gov). Ниже указана структура праймеров и флуоресцентно меченого зонда, используемых в работе:
Rd1 5’-CCGGATGTGGGGACCCTCTTCCTCTAGC-3’, Rd2 5’-GCCATGTCGGGGGAGCCTGAGCTCA-3’ для RASSF1(804); PK51 5' GAA CCG TGT TTC CCT AGA ACC CAG TC 3' и PK387 5' CGG TCC GGC TGT CCT CCT CAC 3' для DAPK1; RASSF2d 5ʹ GCGAGAGAAAAGAGAGGACAGCGGACGAGC 3ʹ RASSF2r 5ʹ CAGCCGGGGTAGGGACCATCGTGGA 3ʹ SFRP1d 5ʹ CCGCCCTGGTCTCTCTCC 3ʹ SFRP1r 5ʹ GCCATGGTCTCGTGCTCC 3ʹ HS3ST2d 5ʹ GCTGTGTTTCTGGGAGGGGTA 3ʹ HS3ST2r 5ʹ GCACAGGAAGCTGTAACACAGGTA 3ʹ TWIST1d 5ʹ CGAGAGAGCAGGCCGGGACGCAA 3ʹ TWIST1r 5ʹ GCCGCCTCCGACGCCCCCA 3ʹ RASSF1md 5' CGGCCAGGGACCAGCTGC 3'(18) RASSF1mz 5' FAM-AGCTCAGGCTCCCCCGACATGGC-BHQ1 3'(23) RASSF1mr 5' GCGAAGCACGGGCCCAAC 3'(18)
ПЦР-продукты разделялись путем электрофореза в 1,2 % агарозе “Low EEO, Type 1-A” (“Sigma”, США) в трис-ацетатном буфере (40 mM Трис-ацетат, pH 8,0; 1 mM ЭДТА). После проведения электрофореза ДНК визуализировали бромистым этидием и фотографировали в УФ-свете. Для оценки длин фрагментов использовали ДНК-маркер “100 bp”.
По завершении ПЦР для RASSF1(173), при помощи программного обеспечения амплификатора “Bio-Rad CFX Manager v.2.1”, устанавливали среднее значение Cq и значение среднеквадратичного отклонения для анализируемых образцов и полученные данные заносили в таблицу.
Результаты и Обсуждение
На рисунке 1 представлены результаты GlaI-ПЦР анализа регуляторной области ген DAPK1 в ДНК клеточных линий Raji, U937 и L68.
Как видно из рисунка, фермент GlaI расщепляет данный фрагмент ДНК в случае клеточной линии Raji и не расщепляет в случае L68 и U937.
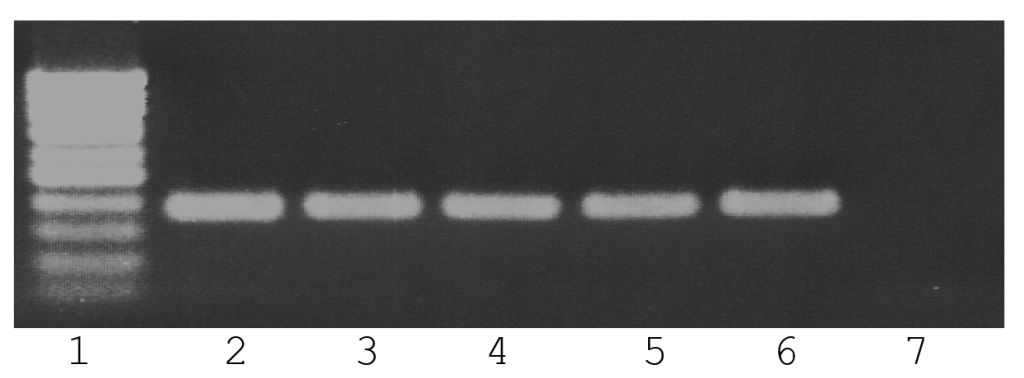
На рисунке 2 представлены результаты GlaI-ПЦР анализа регуляторной области гена HS3ST2 в ДНК клеточных линий Raji, U937 и L68.
Как видно из рисунка, фермент GlaI расщепляет регуляторную область гена HS3ST2 в ДНК Raji и U-937, но не ДНК L68.
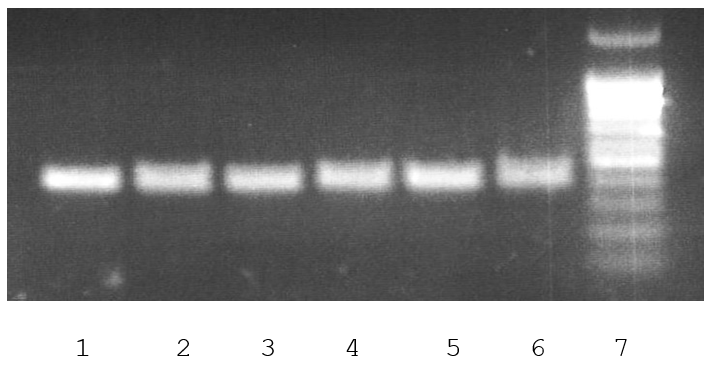
На рисунке 3 представлены результаты GlaI-ПЦР анализа регуляторных областей гена RASSF2 в ДНК клеточных линий Raji, U937 и L68.
Как видно из рисунка, фермент GlaI не расщепляет регуляторную область гена RASSF2 в ДНК Raji, U-937 и L68.
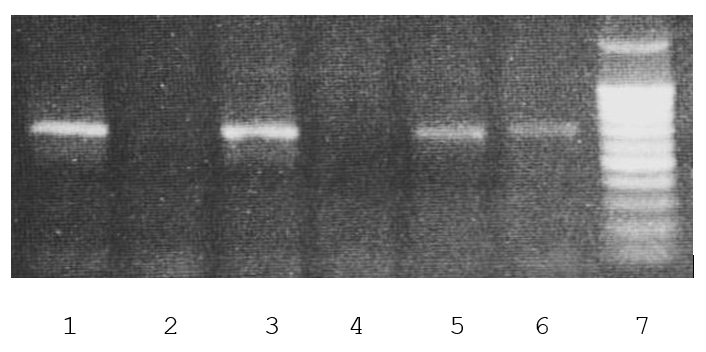
На рисунке 4 представлены результаты GlaI-ПЦР анализа регуляторной области гена TWIST1 в ДНК клеточных линий Raji, U937 и L68
Как видно из рисунка, GlaI расщепляет регуляторную область гена TWIST1 в ДНК Raji и U-937 и не расщепляет в ДНК L68.
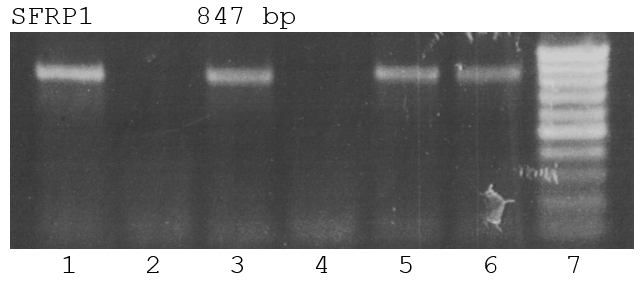
На рисунке 5 представлены результаты GlaI-ПЦР анализа регуляторной области гена SFRP1 в ДНК клеточных линий Raji, U937 и L68.
Как видно из рисунка 5, также как в случае гена TWIST1, GlaI расщепляет регуляторную область гена TWIST1 в ДНК Raji и U-937 и не расщепляет в ДНК L68.
В таблице 1 представлены полученные результаты GlaI-ПЦР анализа изучаемых регуляторных областях генов-онкосупрессоров MGMT, RASSF2, RARB, RASSF1, SFRP1, CEBPD, HS3ST2, TWIST1, SEPT9, IGFBP3 и DAPK1 в ДНК клеточных линий L68, U937 и Raji, а также данные по количеству сайтов RCGY в изучаемых регуляторных областях этих генов (в столбце 3 указана позиция в геноме анализируемого фрагмента ДНК, в столбце 4 указано количество сайтов RCGY в данном фрагменте). В таблице также указано количество расщеплений (ридов) в анализируемых фрагментах, полученных на ДНК L68 (столбик 5), Raji (столбик 6) и U937 (столбик 7) по данным NGS, представленным в работе [2]. При этом 7 и менее сайтов во фрагменте принимаются как фоновое значение и фрагмент с таким числом сайтов считается неметилированным. В столбиках 5-7, после числа расщеплений, в скобках стоит знак (+) или (-), означающий, что в этом фрагменте есть сайты R(5mC)GY (+), или их нет (-), по данным GlaI-ПЦР анализа.
Таблица 1
| Ген | Хромосома | Координаты участка | Число сайтов RCGY | L68 | Raji | U937 | Ссылка |
| MGMT | 10 | 129466966–129467321 | 16 | 3 (−) | 2 (−) | 47 (+) | [6] |
| RASSF2 | 20 | 4823170–4823593 | 13 | 0 (−) | 0 (−) | 0 (+) | Рис. 3 |
| RARB | 3 | 25428202–25429054 | 4 | 0 (−) | 38 (+) | 19 (+) | [5] |
| RASSF1 | 3 | 50340359–50341162 | 26 | 21 (−) | 33 (+) | 0 (−) | [6] |
| SFRP1 | 8 | 41308897–41309743 | 28 | 1 (−) | 97 (+) | 429 (+) | Рис. 5 |
| CEBPD | 8 | 47738031–47738470 | 19 | 2 (−) | 68 (+) | 104 (+) | [6] |
| HS3ST2 | 16 | 22814027–22814727 | 29 | 5 (−) | 54 (+) | 25 (+) | Рис. 2 |
| TWIST1 | 7 | 19117133–19118334 | 28 | 7 (−) | 141 (+) | 148 (+) | Рис. 4 |
| SEPT9 | 17 | 77372935–77373873 | 33 | 2 (−) | 119 (+) | 111 (+) | [6] |
| IGFBP3 | 7 | 45920650–45921466 | 41 | 0 (−) | 32 (+) | 76 (+) | [6] |
| DAPK1 | 9 | 87497541–87497897 | 12 | 0 (−) | 73 (+) | 0 (−) | Рис. 1 |
Как видно из результатов определения статуса метилирования по данным GlaI-ПЦР анализа в ДНК L-68 не метилирован ни один из 11 изучаемых фрагментов. В то же время в ДНК Raji метилированы регуляторные участки всех генов, кроме MGMT и RASSF2, а в ДНК U-937 метилированы все гены, кроме регуляторных участков генов DAPK1, RASSF1 и RASSF2. Таким образом, в ДНК клеточных линий лимфом (Raji – лимфома Беркита и U937 – гистиоцитарная лимфома, соответственно) большинство фрагментов метилировано при отсутствии метилирования всех фрагментов в ДНК L68. При этом, ген RASSF2 не метилрован во всех 3-х ДНК. В опухолевых клеточных линиях, помимо гена RASSF2, ген MGMT не метилирован в ДНК Raji, а гены DAPK1 и RASSF1 – в ДНК U937.
Таким образом, ДНК клеточных линий лимфом Raji и U937 отличаются по метилированию регуляторных участков трех генов – DAPK1, MGMT и RASSF1.
Результаты NGS GlaI-гидролизатов ДНК клеточных Raji, U937 и L68 также представленны в таблице 1. В ДНК Raji и U937 большинство фрагментов (9 и 8, соответственно) содержит метилированные сайты. В ДНК Raji число сайтов R(5mC)GY ниже фона в регуляторных участках генов MGMT и RASSF2, а в ДНК U937 – в регуляторных участков генов RASSF1, RASSF2 и DAPK1 – эти участки не метилированы по данным NGS.
Таким образом, результаты NGS GlaI-гидролизатов ДНК и данные GlaI-ПЦР анализа совпали по всем фрагментам за одним исключением. В ДНК L68 регуляторный участок гена RASSF1 содержит 21 сайт R(5mC)GY, тогда как по данным GlaI-ПЦР анализа этот фрагмент не содержит сайтов R(5mC)GY.
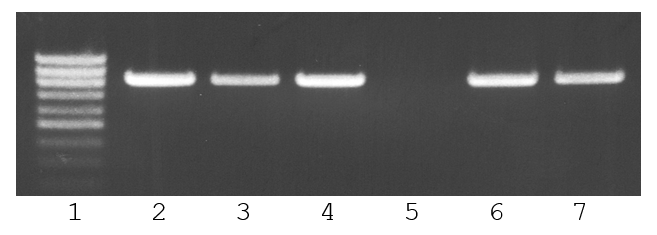
На рис.6 представлены результаты GlaI-ПЦР анализа фрагмента гена-онкосупрессора RASSF1A при более высоком, чем ранее, количестве ДНК клеточных линий L68, U937 и Raji (40 нг). Как видно из рисунка, после расщепления GlaI интенсивность полосы ДНК L68 уменьшается, что может объясняться расщеплением ДНК ферментом GlaI только в одном аллеле диплоидной ДНК (вследствие его метилирования и отсутствия метилирования во втором аллеле). В таком случае NGS GlaI-гидролизатов ДНК будет показывать наличие сайтов R(5mC)GY в данном фрагменте (так как они присутствуют в одном аллее), тогда как GlaI-ПЦР анализ будет показывать отсутствие этих сайтов и неметилированный статус регуляторного участка гена RASSF1A (так как они отсутствуют во втором аллеле и происходит наработка ампликона).
Рисунок 6
Результаты GlaI-ПЦР анализа фрагмента гена-онкосупрессора RASSF1A при более высоком, чем ранее, количестве ДНК клеточных линий
Дорожки:
1- маркер 100 bp,
2- ДНК L68,
3- ДНК L68 + GlaI,
4- ДНК Raji,
5- ДНК Raji + GlaI,
6- ДНК U937,
7- ДНК U937 + GlaI.
Амплификацию проводили в 30 мкл в приборе “Терцик” (“ДНК-Технология”) по следующему протоколу: при 95°C — 3 мин; при 96°C — 30 сек; 29 циклов: 95°C — 10 сек, 68°C — 15 сек, 72°C — 50 сек, 75°C — 5 сек;
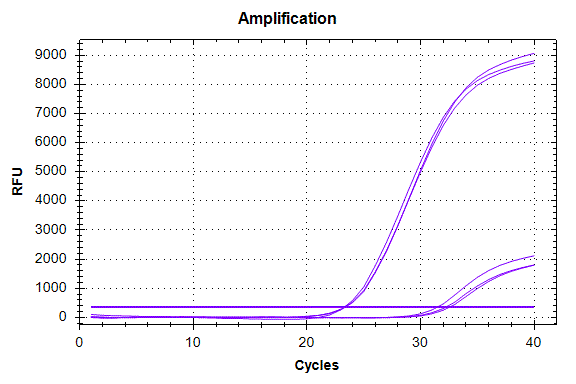
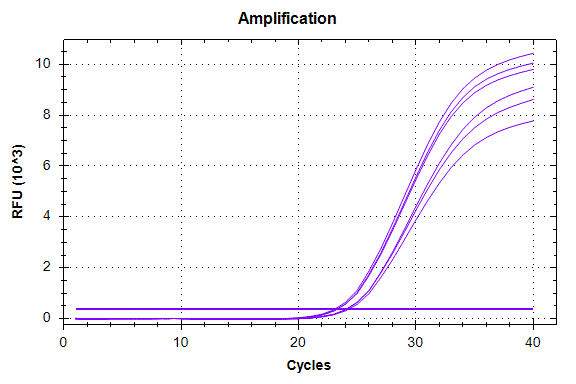
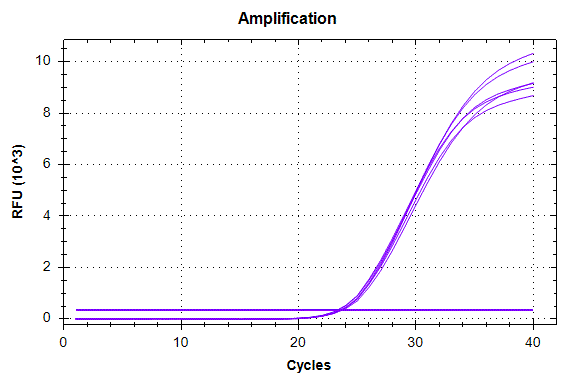
Для подтверждения аллельного метилирования регуляторного участка гена RASSF1A мы провели GlaI-ПЦР анализ в реальном времени более короткого фрагмента (173 нуклеотида) данного участка ДНК, структура которого представлена ниже. В таблице 2 и на рис. 7, 8, 9 представлены данные GlaI-ПЦР анализа этого фрагмента на ДНК клеточных культур L68, U937 и Raji.
ПЦР в реальном времени проводили в термоциклере BIO-RAD CFX по следующему протоколу: при 95°C — 3 мин; при 96°C — 30 сек; 5 «слепых» циклов: 96°C — 10 сек, 64°C (с понижением на 0.4°C в каждом последующем цикле) — 15 сек, 72°C — 10 сек, 75°C — 5 сек; 40 циклов: 96°C — 10 сек, 62°C — 15 сек (с детекцией флуоресцентного сигнала в канале FAM), 72°C — 10 сек, 75°C — 5 сек;
Таблица 2
| DNA | Raji | Raji + GlaI | L68 | L68 + GlaI | U937 | U937 + GlaI |
| Cq | 23,25±0,05 | 32,00±0,55 | 23,27±0,11 | 24,21±0,10 | 23,35±0,16 | 23,55±0,10 |
Как видно из рисунков 7, 8 и 9 в случае U937 значения Cq для ДНК до и после обработки ферментом GlaI отличаются незначительно, в случае ДНК L68 различие составляет около 1 и в случае ДНК Raji оно составляет 8,75. Ранее нами проводилось изучение аллельного метилирования ряда сайтов RCGY в геноме человека и было показано достоверное определение аллельного метилирования методом GlaI-ПЦР анализа [8-10].
Полученные данные GlaI-ПЦР анализа в реальном времени соответствуют метилированию обоих аллелей фрагмента 173 bp в случае ДНК Raji, метилированию одного аллеля в случае ДНК L68 и отсутствию метилирования фрагмента в случае ДНК U937. Таким образом, данные GlaI-ПЦР анализа регуляторного участка гена RASSF1A соответствуют результатам NGS анализа при учете аллельного метилирования данного фрагмента. Существенно меньшее (по данным NGS) число сайтов R(5mC)GY в регуляторном участке гена RASSF1A в случае ДНК L68 по сравнению с ДНК Raji также соответствует метилированию только одного аллеля в ДНК L68.
В итоге можно заключить, что результаты, полученные методами NGS и GlaI-ПЦР анализа соответствуют и дополняют друг друга.
Дополнительно мы установили полученные в NGS числа расщеплений на участках генома, которые анализировались в ранее опубликованных работах по онкогематологическим заболеваниям методами бисульфитного секвенирования (BS-seq) и COBRA. Для всех этих случаев было установлено, что в малигнантных клетках происходит гиперметилирование данных участков по сравнению с нормой.
Таблица 3
| Ген | Хромосома | Позиции участка | Метод анализа метилирования | Ссылка | Число сайтов RCGY | L68 | Raji | U937 |
| FZD8 | 10 | 35641317–35641638 | BS-seq | [11] | 9 | 0 | 128 | 124 |
| TJP1 (ZO-1) | 15 | 29822517–29822687 | BS-seq | [12] | 1 | 2 | 102 | 254 |
| DLC1 | 8 | 13132965–13133256 | BS-seq | [13] | 8 | 0 | 28 | 88 |
| SMAD1 | 4 | 145481539–145481824 | BS-seq | [14] | 11 | 0 | 12 | 40 |
| HOXA4 | 7 | 27130859–27131134 | COBRA | [15] | 13 | 1 | 128 | 210 |
| TERT | 5 | 1294882–1295708 | BS-seq | [16] | 30 | 23 | 68 | 162 |
Как видно из таблицы, полученные при NGS GlaI-фрагментов данные показывают значительное превышение количества сайтов R(5mC)GY в ДНК опухолевых линий по сравнению с ДНК здоровых клеток (L68). Единственной областью, где наблюдается метилирование в контрольных клетках L68, является промоторный участок гена TERT, но и в этом случае число расщеплений в нем по данным NGS значительно уступает соответствующим числам для малигнантных клеток Raji и U937.
Очевидно, что использованный метод определения метилированных сайтов RCGY путем проведения массового параллельного секвенирования GlaI-фрагментов на Illumina имеет ряд недостатков. Он не позволяет детектировать расщепления близко расположенных или сильно отдаленных метилированных сайтов, что связано с необходимостью выбирать лишь фрагменты подходящего для NGS размера (в описанных работах по секвенированию геномов трех клеточных линий изучались фрагменты длиной 140-400 п.н.). Тем не менее, сравнение данных GlaI-ПЦР-анализа с результатами NGS показывает, что их результаты полностью коррелируют. Таким образом, мы предполагаем, что NGS ДНК-фрагментов, являющихся гидролизатами метилзависимой ДНК-эндонуклеазы GlaI, проведенное для геномов больных и здоровых, позволит путем сравнения выявить участки ДНК, аномально метилированные/деметилированные при различных заболеваниях.
Литература
-
Tarasova G.V., Nayakshina T.N., Degtyarev S.Kh.
Substrate specificity of a new methyl-directed DNA endonuclease GlaI.
BMC Molecular Biology. 2008; 9:7.
https://doi.org/10.1186/1471-2199-9-7 -
Abdurashitov M.A., Tomilov V.N., Gonchar D.A., Kuznetsov V.V., Degtyarev S.Kh.
Mapping of R(5mC)GY sites in the genome of human malignant cell line Raji.
Biology and Medicine (Aligarh). 2015; 7(4): BM-135-15. -
Абдурашитов М.А., Томилов В.Н., Гончар Д.А., Снежкина А.В., Краснов Г.С., Кудрявцева А.В., Дегтярев С.Х.
Сравнительный анализ метилирования сайтов RCGY в геномах клеточных линий L-68, Raji и U-937.
Эпигенетическая ДНК диагностика. 2019; 2019(1). -
Genome Reference Consortium.
Human genome assembly GRCh38.
https://www.ncbi.nlm.nih.gov/assembly/GCF_000001405.39 -
Hernando-Herraez I., Garcia-Perez R., Sharp A.J., Marques-Bonet T.
DNA methylation: insights into human evolution.
PLoS Genetics. 2015; 11(12): e1005661.
https://doi.org/10.1371/journal.pgen.1005661 -
Гончар Д.А., Акишев А.Г., Дегтярев С.Х.
BlsI- и GlaI-ПЦР-анализ — новый метод исследования метилированных участков ДНК.
Вестник биотехнологии и физико-химической биологии имени Ю.А. Овчинникова. 2010; 6(1): 5–12.
http://sciencerussian.sibenzyme.com/papers/epigenetics/blsi-i-glai-ptsr-analiz-novyj-metod-issledovaniya-metilirovannykh-uchastkov-dnk -
Акишев А.Г., Гончар Д.А., Абдурашитов М.А., Дегтярев С.Х.
Эпигенетическое типирование малигнантных клеточных линий человека с помощью BlsI- и GlaI-ПЦР-анализа.
Вестник биотехнологии и физико-химической биологии имени Ю.А. Овчинникова. 2011; 7(3): 5–16.
http://sciencerussian.sibenzyme.com/papers/epigenetics/epigeneticheskoe-tipirovanie-malignantnykh-kletochnykh-linij-cheloveka-s-pomoshchyu-blsi-i-glai-ptsr-analiza -
Абдурашитов М.А., Куксова А.Н., Акишев А.Г., Землянская Е.В., Дегтярев С.Х.
MteI-ПЦР-анализ — новый метод определения статуса метилирования GC-богатых регуляторных участков генов-онкосупрессоров человека.
Вестник биотехнологии и физико-химической биологии имени Ю.А. Овчинникова. 2013; 9(3): 15–23.
http://sciencerussian.sibenzyme.com/papers/epigenetics/mte-pcr-analysis -
Акишев А.Г., Нетесова Н.А., Абдурашитов М.А., Дегтярев С.Х.
Определение полиморфизма 5mC/T в повторе AluSx (Chr16:75033884) в препаратах ДНК из крови человека методами GlaI- и FatI-ПЦР-анализа.
Эпигенетическая ДНК диагностика. 2021; 1: 1–12.
https://doi.org/10.26213/SE.2021.11.76.001 -
Акишев А.Г., Нетесова Н.А., Абдурашитов М.А., Дегтярев С.Х.
Определение полиморфизма 5mC/T в позиции Chr1:245618129 в препаратах ДНК из крови человека методами GlaI- и FatI-ПЦР-анализа.
Эпигенетическая ДНК диагностика. 2021; 1: 13–23.
https://doi.org/10.26213/SE.2019.69.42836 -
Акишев А.Г., Нетесова Н.А., Абдурашитов М.А., Дегтярев С.Х.
Определение полиморфизма G/C в позиции Chr20:37352001 в препаратах ДНК из крови человека методами GlaI- и Bst2UI-ПЦР-анализа.
Генетика. 2023; 59(11): 1303–1312.
https://doi.org/10.31857/S0016675823110012 -
Bethge N., Honne H., Hilden V., Trøen G., Eknæs M., Liestøl K., et al.
Identification of highly methylated genes across various types of B-cell non-Hodgkin lymphoma.
PLoS ONE. 2013; 8(11): e79602.
https://doi.org/10.1371/journal.pone.0079602 -
Cen J., Shen J., Wang X., Kang H., Wang L., Sun L., Yu L.
Association between lymphoma prognosis and aberrant methylation of ID4 and ZO-1 in bone marrow and paraffin-embedded lymphoma tissues of treatment-naive patients.
Oncology Reports. 2013; 30: 455–461.
https://doi.org/10.3892/or.2013.2450 -
Ullmannova-Benson V., Guan M., Zhou X., Tripathi V., Yang X.Y., Zimonjic D.B., Popescu N.C.
DLC1 tumor suppressor gene inhibits migration and invasion of multiple myeloma cells through the RhoA GTPase pathway.
Leukemia. 2009; 23(2): 383–390.
https://doi.org/10.1038/leu.2008.285 -
Stelling A., Wu C.T., Bertram K., Hashwah H., Theocharides A., Manz M.G., Tzankov A., Müller A.
Pharmacological DNA demethylation restores SMAD1 expression and tumor suppressive signaling in diffuse large B-cell lymphoma.
Blood Advances. 2019; 3(20): 3020–3032.
https://doi.org/10.1182/bloodadvances.2019000210 -
Strathdee G., Holyoake T.L., Sim A., Parker A., Oscier D.G., Melo J.V., et al.
Inactivation of HOXA genes by hypermethylation in myeloid and lymphoid malignancy is frequent and associated with poor prognosis.
Clinical Cancer Research. 2007; 13(17): 5048–5055.
https://doi.org/10.1158/1078-0432.CCR-07-0919 -
Chebly A., Ropio J., Peloponese J.M., Poglio S., Prochazkova-Carlotti M., Cherrier F., et al.
Exploring hTERT promoter methylation in cutaneous T-cell lymphomas.
Molecular Oncology. 2021.
https://doi.org/10.1002/1878-0261.12946